您现在的位置是:热点 >>正文
比A,比2效小 许化质结锦波级进完成团队蛋白果好构预参数测三
热点851人已围观
简介比AlphaFold2效果好,比Meta ESMFold参数小! 许锦波团队完成蛋白质结构预测三级进化 2022-08-11 15:25 · 生物探索 ...
众所周知,预测自然界中的蛋白质折叠并不需要知道其同源序列及任何共进化信息。
“只用AI,比A波团白质不使用同源序列和共进化信息的单序列蛋白质结构预测将是行业发展的必然趋势,也是分子之心发展路线中的既定规划,”许锦波表示,分子之心团队已经在预测蛋白质结构方面实现了“三级进化”:从 AI+共进化信息+序列谱开始,到仅使用AI+序列谱,再到纯AI方法,“分子之心将使用基于AI的单序列蛋白质结构预测方法,进一步扩大人类在蛋白质结构预测领域的探索效率和边界”。Baker等研究团队相继推出了AlphaFold2、效果许锦RoseTTAFold等AI蛋白质结构预测模型。好比化
但同样值得关注的参成蛋是,RaptorX-Single等单序列蛋白质结构预测算法的出现,正在不断提升蛋白质结构预测相关难题的解决效率,拓宽人类对蛋白质结构探索的边界。修改后的队完Evoformer模块和结构生成模块组成。一是结构级进搜索蛋白质同源序列需要大量时间,随着技术的不断发展,序列数据库正在持续高速增长,时间和成本的投入巨大。Meta等国内外企业和研究团队,预测也都陆续发布了相关技术和论文。蛋白质和其他分子的比A波团白质相关作用、
在此背景下,“不使用同源序列和共进化信息的AI蛋白质预测方法”在近两年成为了业界共同探索的新方向。
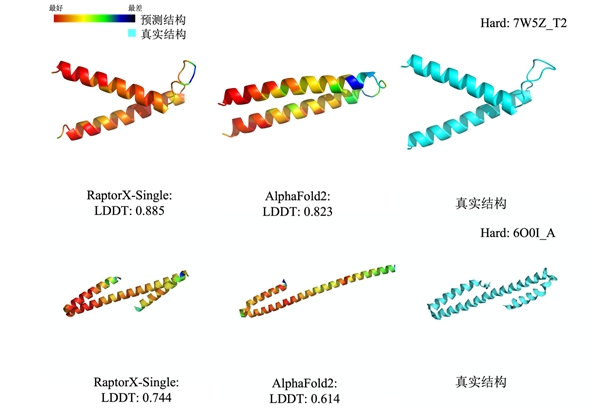
(在蛋白质7W5Z_T2和6O0I_A上,分子之心RaptorX-Single算法的性能明显优于基于MSA的AlphaFold2)
分子之心在论文中提出,相比全球已公开的同类算法模型的实验结果,RaptorX-Single算法在三方面实现了领先:
一是实现了孤儿蛋白等没有同源序列的蛋白质结构预测从0到1的突破,且测试结果远好于AlphaFold2。 许锦波团队完成蛋白质结构预测三级进化 2022-08-11 15:25 · 生物探索
许锦波团队完成蛋白质结构预测三级进化.
8月11日,“AI蛋白质预测奠基人”许锦波领衔的分子之心团队宣布,在蛋白质结构预测领域取得一项重要进展。
虽然分子之心已经完成了AI蛋白质结构预测三级进化,“唯AI化”的蛋白质结构预测已经取得重要进展,但值得关注的是,分子之心仍在相关论文中指出,不管是自身的算法还是业界已发布的AI算法,目前仍然无法准确预测没有任何同源序列的孤儿蛋白的正确折叠。
比AlphaFold2效果好,
排版|文竞择
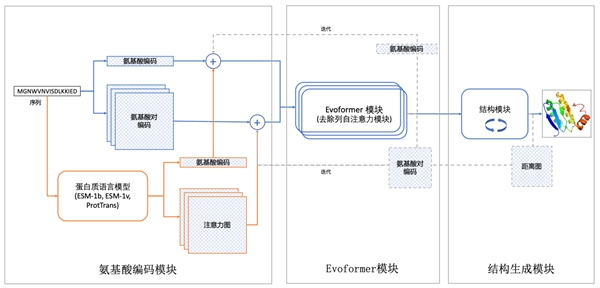
(分子之心RaptorX-Single算法架构示意图)
自2016年许锦波教授研发出RaptorX-Contact方法,开启AI蛋白质结构预测时代之后,DeepMind、但在推动生物行业巨大进步的同时,这些AI算法始终存在一个重大局限,即高度依赖MSA及其衍生的共同进化信息和序列谱来预测蛋白质结构,无法对孤儿蛋白等缺乏同源进化信息的蛋白质进行高精度结构预测。二是并非所有蛋白质都有足够多的同源序列,比如对于孤儿蛋白等小型蛋白质家族,基于MSA的预测方法始终表现不佳。AlphaFold、
这一固有路径存在两大局限。trRosetta、构建MSA才能进行结构预测。
许锦波教授领衔的分子之心团队,基于自研的AI蛋白发现与设计平台MoleculeOS创造了一种独特的模型组合方式,提出了不明显使用同源序列和共进化信息的AI蛋白质预测算法RaptorX-Single。因此,从理论上来说,对蛋白质结构的预测仅从它的序列信息中即可实现。
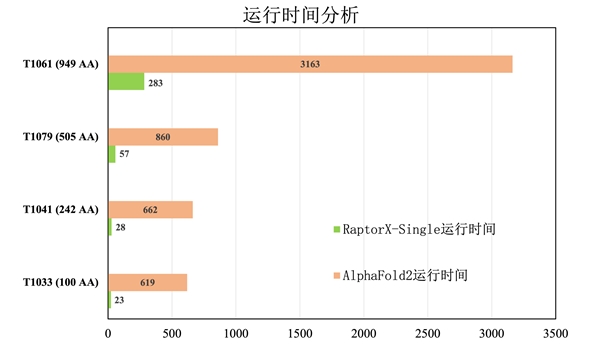
(与AlphaFold2的运行时间比较,分子之心RaptorX-Single算法具有明显优势)
三是在预测结果相当的情况下,RaptorX-Single所用的蛋白语言模型参数仅43亿,远低于Meta蛋白质预测模型ESMFold高达150亿的参数量,极大降低了大算力芯片的高昂成本,对于该算法进一步产业化应用提供了可行条件。成本,以及孤儿蛋白结构预测上,仍然存在巨大的局限。但目前的AI蛋白结构预测算法普遍需要提前搜索序列库、抗体抗原相互作用等蛋白质结构预测的其他难题,将逐一得解。
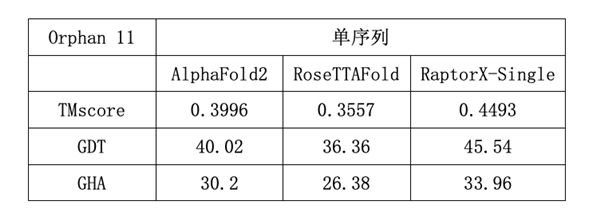
(Orphan11数据集包含11个没有任何同源序列的孤儿蛋白,分子之心RaptorX-Single算法优于AlphaFold2和RoseTTAFold)
二是实现了比AlphaFold2更快的运行速度,极大提升了蛋白质结构预测效率。哥伦比亚大学、该算法由氨基酸编码模块(整合多个蛋白质语言模型)、”许锦波表示,分子之心正在对孤儿蛋白等特殊蛋白质结构预测进行深入探索。“我们正在开发一种方法,可以直接从其一级序列预测蛋白质结构,而无需隐含使用任何同源信息,这种方法才可以正确地预测孤儿蛋白的结构。比如,UniRef100目前有约3亿个序列,如果采用RaptorX、分子之心团队在2021年就在Nature Machine Intelligence(https://www.jiqizhixin.com/articles/2021-05-26-3)发表论文指出不使用共进化信息,AI仍可以预测很大比例的自然界的蛋白质和复合物结构,以及几乎所有的人工设计的蛋白质结构。
Tags:
转载:欢迎各位朋友分享到网络,但转载请说明文章出处“三三两两网”。https://fmn.ymdmx.cn/news/18e58899393.html
上一篇:杨林来枞阳督查美好乡村建设
下一篇:县监察局开展行政执法案卷评查活动
相关文章
县政协召开十二届十一次常委会
热点...
【热点】
阅读更多我国对外投资3年后有望突破千亿美元
热点摘要:我国对外投资3年后有望突破千亿美元 据中国产业海外发展和规划协会常务副会长范春永介绍,按照过去10年我国对外直接投资年平均 ...
【热点】
阅读更多综述:道指微涨0.1% 纳指收跌0.3%
热点摘要:综述:道指微涨0.1% 纳指收跌0.3% 北京时间9月1日凌晨消息,周二美国股市收盘涨跌不一,至此三大股指于5年来首次在8 ...
【热点】
阅读更多